Analisi dati
La quantificazione è il passaggio che trasforma un insieme di spettri NMR in una matrice di dati. Questo obiettivo può essere raggiunto seguendo due percorsi complementari: risolvendo l’insieme dei segnali assegnando ciascuno (o la maggior parte) al corretto metabolita - ciò implica che noi assumiamo di sapere a priori quali molecole possono essere presenti nella miscela - e misurando le intensità degli specifici picchi di interesse (targeted analysis), oppure elaborando gli spettri "tal quali" indipendentemente da qualsiasi considerazione e basandosi sulle strutture di correlazione incorporate nello spettro 1H 1D grazie alla sua ridondanza intrinseca (pattern recognition) .
Quest'ultima strategia viene applicata all'intero insieme di spettri contemporaneamente e richiede una serie di passaggi automatici o semi-automatici che possono includere l'allineamento dei segnali e successivamente la discretizzazione degli spettri suddividendo l'intervallo spettrale in piccole frazioni (bucketing o binning). Il risultato è una matrice di dati che incorpora tutte le informazioni incluse negli spettri, ma le cui variabili (intervalli spettrali) non hanno un significato metabolico diretto: si effettua quindi l'analisi multivariata per svelare le relazioni strutturali e metaboliche sottese agli spettri. 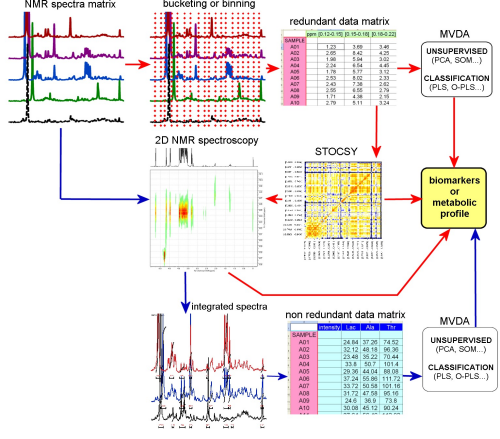
Sia la targeted analysis che la pattern recognition richiedono l'assegnazione dei segnali di risonanza ai loro metaboliti di origine. Questa procedura richiede l'abilità dell'operatore, che si può basare su una vasta gamma di tecniche 2D NMR, (spettroscopia omonucleare bidimensionale, come la spettroscopia di correlazione (COrrelation SpectroscopY - COSY) e spettroscopia di correlazione totale (TOtal Correlation SpectroscopY - TOCSY) o esperimenti di spettroscopia eteronucleare bidimensionale come la spettroscopia eteronucleare a coerenza di singolo quanto (Heteronuclear Single Quantum Coherence - HSQC) o la spettroscopia eteronucleare di correlazione di legame multiplo (Heteronuclear Multiple Bond Correlation - HMBC)), su spettri di riferimento, su dati di letteratura.
Un altro strumento disponibile è dato dalla spettroscopia statistica di correlazione totale (Statistical TOtal Correlation SpectroscopY - STOCSY): in questo metodo le variabili della matrice sono correlate in coppia e i coefficienti di correlazione sono disposti in una matrice di correlazione. Le correlazioni più forti sono dovute a segnali appartenenti alle stesse molecole.
Le variabili della matrice di dati prodotta dall'analisi supervised sono le concentrazioni reali dei metaboliti rilevati e l'applicazione di metodi multivariati può portare direttamente alla comprensione del sistema metabolico oggetto di studio.
Sia la targeted analysis che la pattern recognition contribuiscono a fornire i profili metabolici del sistema e a rilevare i biomarcatori che caratterizzano i processi biologici che si verificano in esso.
Dopo questi passaggi, vengono applicate le tecniche multivariate per il rilevamento - in modo unsupervised per individuare correlazioni intrinseche nel campo dati e/o la presenza di valori anomali (comunemente mediante Analisi in Componenti Principali, PCA) o supervised utilizzando variabili esterne note (come con Partial Least Squares Analysis, PLS, Orthogonal-PLS, Multiple Correspondence Analysis, MCA) o per classificazione (PLS, SIMCA, Multiway analysis ecc.)
